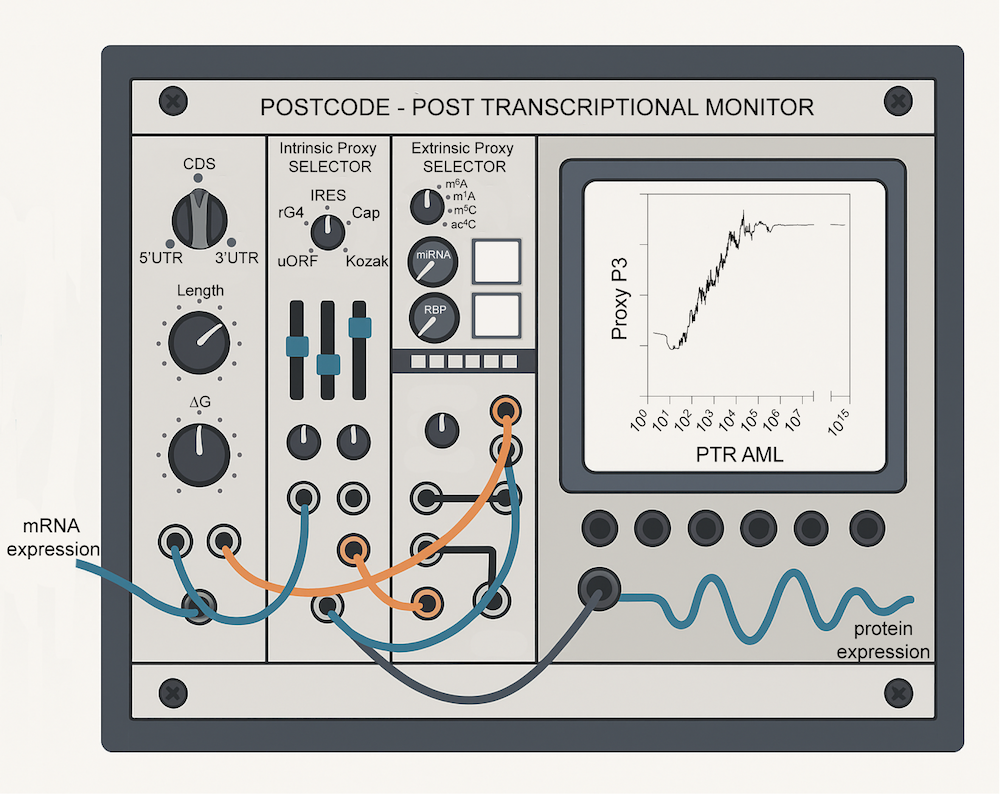
Une étude de l’Institut Cochin publiée dans Communications Biology (Nature Portfolio) apporte un nouvel éclairage sur la régulation post‑transcriptionnelle de l’expression des gènes dans la leucémie aiguë myéloïde (LAM). En combinant des données transcriptomiques et protéomiques issues de patients atteints de LAM, les auteurs montrent que le ratio protéine/ARNm (PTR) constitue un indicateur robuste et conservé des régulations post-transcriptionnelles chez ces patients. L’étude identifie les déterminants moléculaires majeurs contrôlant la traduction, la stabilité et la localisation des ARNm, et introduit un nouvel outil, POSTCODE, pour analyser ces mécanismes dans tout type de modèle et à grande échelle.
© Institut Cochin
Mise en évidence d’une régulation post-transcriptionnelle conservée dans la LAM
L’expression des gènes ne se limite pas à la transcription : de nombreux mécanismes post‑transcriptionnels contrôlent la traduction des ARNm en protéines, leur stabilité ou leur séquestration cellulaire. Dans la leucémie aiguë myéloïde (LAM), ces processus restent encore mal caractérisés à l’échelle globale.
Dans cette étude, les chercheurs de l’Institut Cochin, en collaboration avec plusieurs partenaires nationaux et internationaux ont intégré des données transcriptomiques et protéomiques provenant de 44 patients atteints de LAM afin de calculer les ratios protéine/ARNm (PTR), un paramètre synthétique reflétant l’ensemble des régulations post‑transcriptionnelles. Ils montrent que ces PTR sont remarquablement conservés entre patients, sous‑types de LAM et même entre différents tissus humains, suggérant l’existence de mécanismes fondamentaux et universels.
Déterminants moléculaires des PTR et développement de l’outil POSTCODE
L’analyse détaillée de plus d’un millier de « proxys » (caractéristiques) moléculaires — liés aux régions 5’UTR, CDS et 3’UTR des ARNm, ainsi qu’aux propriétés des protéines — permet d’identifier les déterminants clés de ces PTR. Ces déterminants incluent notamment l’efficacité de l’initiation et de l’élongation de la traduction, la composition en codons, la stabilité des ARNm et leur localisation subcellulaire.
Enfin, les auteurs développent POSTCODE, un outil bioinformatique destiné à annoter des jeux de données « omiques » (transcriptomique, protéomique, ribosome profiling) à partir de ces proxys. POSTCODE permet ainsi de révéler des régulations post-transcriptionnelles jusque-là invisibles dans les analyses classiques, ouvrant de nouvelles perspectives pour la compréhension de la LAM et l’identification de stratégies de médecine de précision.
POSTCODE est proposé sous la forme d’une application web user‑friendly, utilisable sans aucune compétence en programmation, et applicable à tout type de problématique biologique humaine, au-delà du seul cadre de la LAM. L’outil est librement accessible à l’adresse suivante : https://postcode-lab.shinyapps.io/POSTCODE/.
À lire aussi

Cochrane France : un engagement renforcé pour la santé fondée sur les preuves
Centre francophone du réseau international Cochrane, Cochrane France œuvre au développement et à la diffusion des données probantes pour éclairer les décisions en santé. Placé sous la direction du Pr Isabelle Boutron, le centre poursuit son engagement scientifique...

Summers Schools 2026 : une expérience internationale d’excellence à l’UFR de Médecine
Cet été l’UFR de Médecine de l’Université Paris Cité ouvrira pour la première fois à des étudiants du monde entier des programmes intensifs d’apprentissage et d’échanges, organisés entièrement en anglais et ouverts à une audience internationale. Pendant deux semaines,...
![[Keynote recherche] Florence Niedergang – Macrophages, virus et bactéries : qui manipule qui ?](https://u-paris.fr/sante/wp-content/uploads/sites/19/2026/02/3d-abstract-medical-background-with-close-up-virus-cells1.jpg)
[Keynote recherche] Florence Niedergang – Macrophages, virus et bactéries : qui manipule qui ?
Florence Niedergang est directrice de l’Institut Cochin et directrice de recherche au CNRS. Elle dirige l’équipe Biologie des phagocytes, infection et immunité, dont les travaux portent sur les interactions hôte–pathogènes, en particulier les mécanismes...
Vers une école de chirurgie d’excellence : le Campus chirurgical du Grand Paris
Ancré sur le site historique de l’ancien hôpital Broussais dans le 14ᵉ arrondissement, le Campus chirurgical du Grand Paris incarne un tournant majeur pour la formation, la recherche et l’innovation en chirurgie. Cette nouvelle École de Chirurgie, fruit d’une...
